3 Operaciones de datos de atributos
Prerrequisitos
- Este capítulo requiere que se instalen y adjunten los siguientes paquetes:
library(sf) # paquete de datos vectoriales introducido en el capítulo 2
library(terra) # paquete de datos raster introducido en el capítulo 2
library(dplyr) # paquete tidyverse para la manipulación de marcos de datos- También depende de spData, que carga los conjuntos de datos utilizados en los ejemplos de código de este capítulo:
3.1 Introducción
Los datos de atributos son la información no espacial asociada a los datos geográficos (geométricos).
Un ejemplo sencillo es el de una parada de autobús: su posición suele estar representada por coordenadas de latitud y longitud (datos geométricos), además de su nombre.
La parada Elephant & Castle / New Kent Road de Londres, por ejemplo, tiene unas coordenadas de -0,098 grados de longitud y 51,495 grados de latitud que pueden representarse como POINT (-0,098 51,495) en la representación sfc descrita en el capítulo 2.
Los atributos, como el nombre atributo de la función POINT (por utilizar la terminología de Simple Features) son el tema de este capítulo.
Otro ejemplo es el valor de elevación (atributo) de una celda específica de la cuadrícula en los datos raster. A diferencia del modelo de datos vectoriales, el modelo de datos raster almacena la coordenada de la celda de la cuadrícula de forma indirecta, lo cual significa que la distinción entre atributo e información espacial es menos clara. Para ilustrar este punto, piensa en un píxel en la 3ª fila y la 4ª columna de una matriz raster.
Su ubicación espacial está definida por su índice en la matriz: se mueve desde el origen cuatro celdas en la dirección x (normalmente este y derecha en los mapas) y tres celdas en la dirección y (normalmente sur y abajo). La resolución del raster define la distancia para cada paso en x e y que se especifica en un cabezal. La cabecera es un componente vital de los conjuntos de datos raster que especifica cómo se relacionan los píxeles con las coordenadas geográficas (véase también el capítulo ??).
Esto muestra cómo manipular objetos geográficos basados en atributos como los nombres de las paradas de autobús en un conjunto de datos vectoriales y las elevaciones de los píxeles en un conjunto de datos rasterizados. En el caso de los datos vectoriales, esto implica técnicas como crear subconjuntos y o agregaciones (véanse las secciones 3.2.1 y 3.2.3).
Las secciones 3.2.4 y 3.2.5 demuestran cómo unir datos en objetos de características simples utilizando un ID compartido y cómo crear nuevas variables, respectivamente.
Cada una de estas operaciones tiene un equivalente espacial:
el operador [ en R básico, por ejemplo, funciona igualmente para subconjuntar objetos basados en su atributo y objetos espaciales; también se pueden unir atributos en dos conjuntos de datos geográficos utilizando uniones espaciales.
Esto es una buena noticia: las habilidades desarrolladas en este capítulo son transferibles.
El capítulo ?? extiende los métodos presentados aquí al mundo espacial.
Después de una inmersión profunda en varios tipos de operaciones de atributos vectoriales en la siguiente sección, las operaciones de datos de atributos raster se cubren en la Sección (manipulando-objetos-raster), que demuestra cómo crear capas raster que contienen atributos continuos y categóricos y cómo extraer valores de celdas de una o más capas (subconjunto raster). La sección 3.3.2 proporciona una visión general de las operaciones ráster “globales” que pueden utilizarse para resumir conjuntos de datos raster completos.
3.2 Manipulación de atributos vectoriales
Los conjuntos de datos vectoriales geográficos están bien soportados en R gracias a la clase sf, que extiende la clase data.frame de R.
Al igual que los marcos de datos, los objetos sf tienen una columna por variable de atributo (como ‘nombre’) y una fila por observación o característica (por ejemplo, por estación de autobuses).
Los objetos sf se diferencian de los marcos de datos básicos porque tienen una columna de geometría de la clase sfc que puede contener una serie de entidades geográficas (uno o ‘múltiples’ puntos, líneas y polígonos) por fila.
Esto se describió en el capítulo 2, donde se demostró cómo los métodos genéricos como plot() y summary() funcionan con los objetos sf.
sf también proporciona genéricos que permiten que los objetos sf se comporten como marcos de datos normales, como se muestra al imprimir los métodos de la clase:
methods(class = "sf") # métodos para objetos sf, se muestran los 12 primeros
#> [1] aggregate cbind coerce
#> [4] initialize merge plot
#> [7] print rbind [
#> [10] [[<- $<- show Muchos de ellos (aggregate(), cbind(), merge(), rbind() y [) sirven para manejar marcos de datos.
Por ejemplo, rbind() une dos marcos de datos, uno “sobre” el otro.
$<- crea nuevas columnas.
Una característica clave de los objetos sf es que almacenan datos espaciales y no espaciales de la misma manera, como columnas en un data.frame.
La columna de geometría de los objetos sf suele llamarse geometría o geom, pero puede utilizarse cualquier nombre.
El siguiente comando, por ejemplo, crea una columna de geometría llamada g:
st_sf(data.frame(n = world$name_long), g = world$geom)
wkb_geometry y the_geom.
Los objetos sf también pueden extender las clases tidyverse para marcos de datos, tibble y tbl.
.
Por lo tanto, sf permite dar rienda suelta a toda la potencia de las capacidades de análisis de datos de R en los datos geográficos, tanto si se utiliza la base de R como las funciones de tidyverse para el análisis de datos.
(Ver Rdatatable/data.table#2273 para ver la compatibilidad entre los objetos sf y el paquete data.table).
Antes de utilizar estas capacidades, merece la pena repasar cómo descubrir las propiedades básicas de los objetos de datos vectoriales.
Empecemos por utilizar las funciones básicas de R para conocer el conjunto de datos world del paquete spData:
class(world) # es un objeto sf y un marco de datos (ordenado)
#> [1] "sf" "tbl_df" "tbl" "data.frame"
dim(world) # es un objeto bidimensional, con 177 filas y 11 columnas
#> [1] 177 11world contiene diez columnas no geográficas (y una columna que contiene una lista de geometrías) con casi 200 filas que representan los países del mundo.
La función st_drop_geometry() mantiene sólo los datos de los atributos de un objeto sf, es decir, elimina su geometría:
world_df = st_drop_geometry(world)
class(world_df)
#> [1] "tbl_df" "tbl" "data.frame"
ncol(world_df)
#> [1] 10La eliminación de la columna de geometría antes de trabajar con los datos de atributos puede ser útil; los procesos de manipulación de datos pueden ejecutarse más rápido cuando trabajan sólo con los datos de atributos y las columnas de geometría no siempre son necesarias.
En la mayoría de los casos, sin embargo, tiene sentido mantener la columna de geometría, lo que explica que la columna sea “pegajosa” (permanece después de la mayoría de las operaciones de atributos a menos que se elimine específicamente).
Las operaciones de datos no espaciales sobre objetos sf sólo cambian la geometría de un objeto cuando es apropiado (por ejemplo, disolviendo los bordes entre polígonos adyacentes tras una agregación).
Convertirse en un experto en la manipulación de datos de atributos geográficos significa convertirse en un experto en la manipulación de marcos de datos.
Para muchas aplicaciones, el paquete tidyverse dplyr ofrece un enfoque eficaz para trabajar con marcos de datos.
La compatibilidad con Tidyverse es una ventaja de sf frente a su predecesor sp, pero hay que evitar algunos inconvenientes (véase la viñeta complementaria tidyverse-pitfalls en geocompr.github.io para más detalles).
3.2.1 Subconjuntos de atributos vectoriales
Los métodos de subconjunto de R base incluyen el operador [ y la función subset().
Las funciones clave para manejar y crear subconjuntos de dplyr son filter() y slice() para crear subconjuntos de filas, y select() para crear subconjuntos de columnas.
Ambos planteamientos conservan los componentes espaciales de los datos de atributos en los objetos sf, mientras que si se utiliza el operador $ o la función dplyr pull() para devolver una única columna de atributos como vector se perderán los datos de atributos, tal y como veremos.
Esta sección se centra en el subconjunto de marcos de datos sf; para más detalles sobre el subconjunto de vectores y marcos de datos no geográficos recomendamos leer la sección 2.7 de An Introduction to R (R Core Team, Smith, and Team 2021) y el Capítulo 4 de Advanced R Programming (Wickham 2019), respectivamente.
El operador [ puede dividir tanto filas como columnas.
Los índices colocados dentro de los corchetes situados directamente después del nombre de un objeto de marco de datos especifican los elementos que se quieren conservar.
El comando object[i, j] significa ’devolver las filas representadas por i y las columnas representadas por j, donde i y j suelen contener enteros o TRUE y FALSE (los índices también pueden ser caracteres, indicando los nombres de las filas o las columnas).
object[5, 1:3], por ejemplo, significa devolver datos que contengan la 5ª fila y las columnas 1 a 3: el resultado debería ser un marco de datos con sólo 1 fila y 3 columnas, y una cuarta columna de geometría si es un objeto sf.
Si se deja i o j vacía se devuelven todas las filas o columnas, por lo que world[1:5, ] devuelve las cinco primeras filas y las 11 columnas que componen el marco de datos.
Los ejemplos que hay a continuación demuestran la creación de subconjunto con R base.
Adivina el número de filas y columnas de los marcos de datos sf devueltos por cada comando y comprueba los resultados en tu propio ordenador (consulta el final del capítulo para ver más ejercicios):
world[1:6, ] # Subconjunto de las filas 1 a 6
world[, 1:3] # Subconjunto de las columnas 1 a 3
world[1:6, 1:3] # Subconjunto de las filas 1 a 6 y las columnas 1 a 3
world[, c("name_long", "pop")] # Columnas por nombre
world[, c(T, T, F, F, F, F, F, T, T, F, F)] # Selección de columnas por índices lógicos
world[, 888] # Índice representando una columna no existeneteUna demostración de la utilidad de utilizar vectores lógicos para el subconjunto se muestra en el fragmento de código siguiente.
Esto crea un nuevo objeto, small_countries, que contiene las naciones cuya superficie es inferior a 10.000 km2:
i_small = world$area_km2 < 10000
summary(i_small) # un vector lógico
#> Mode FALSE TRUE
#> logical 170 7
small_countries = world[i_small, ]El objeto i_small (abreviatura de “índice” que representa a los países pequeños) es un vector lógico que se puede utilizar para agrupar los siete países más pequeños del mundo por su superficie.
Un comando más conciso, que omita el objeto intermediario (i_small), genera el mismo resultado:
small_countries = world[world$area_km2 < 10000, ]La función base de R subset() proporciona otra forma de conseguir el mismo resultado:
small_countries = subset(world, area_km2 < 10000)Las funciones de R base son maduras, estables y ampliamente utilizadas, lo que las convierte en una opción sólida, especialmente en contextos en los que la reproducibilidad y la fiabilidad son fundamentales.
Las funciones de dplyr permiten flujos de trabajo “ordenados” que algunas personas (incluidos los autores de este libro) encuentran intuitivos y productivos para el análisis interactivo de datos, especialmente cuando se combinan con editores de código como RStudio que permiten autocompletar los nombres de las columnas.
A continuación se muestran las funciones clave para el subconjunto de marcos de datos (incluidos los marcos de datos sf) con las funciones dplyr.
select() selecciona las columnas por nombre o posición.
Por ejemplo, podrías seleccionar sólo dos columnas, name_long y pop, con el siguiente comando:
Nota: al igual que con el comando equivalente en R base (world[, c("name_long", "pop")]), la columna geom permanece.
select() también permite seleccionar un rango de columnas con la ayuda del operador ::
# Selecciona todas las columnas entre name_long y pop (incluidas)
world2 = dplyr::select(world, name_long:pop)Puedes eliminar columnas específicas con el operador -:
# Muestra todas las columnas excepto subregion y area_km2
world3 = dplyr::select(world, -subregion, -area_km2)Crear subconjuntos y renombrar columnas al mismo tiempo con la sintaxis nuevo_nombre = antiguo_nombre:
world4 = dplyr::select(world, name_long, population = pop)Cabe destacar que el comando anterior es más conciso que el equivalente en R base, el cual requiere dos líneas de código:
world5 = world[, c("name_long", "pop")] # subagrupar las columnas por nombre
names(world5)[names(world5) == "pop"] = "population" # renombrar la columna manualmenteselect() también funciona con “funciones de ayuda” para operaciones más avanzadas, como contains(), starts_with() y num_range() (véase la página de ayuda con ?select para más detalles).
La mayoría de los verbos de dplyr devuelven un marco de datos, pero se puede extraer una sola columna como vector con pull().
Puede obtener el mismo resultado en R base con los operadores de subconjunto de listas $ y [[, los tres comandos siguientes devuelven el mismo vector numérico:
pull(world, pop)
world$pop
world[["pop"]]slice() es el equivalente de fila de select().
El siguiente fragmento de código, por ejemplo, selecciona las filas 1 a 6:
slice(world, 1:6)filter() es el equivalente de dplyr a la función subset() de R base.
Mantiene sólo las filas que coinciden según los criterios dados, por ejemplo, sólo los países con un área por debajo de un determinado umbral, o con un promedio alto de esperanza de vida, como se muestra en los siguientes ejemplos:
world7 = filter(world ,area_km2 < 10000) # países con un área menor a 10.000km2
world7 = filter(world, lifeExp > 82) # con una esperanza de vida superior a 82 añosEl conjunto estándar de operadores de comparación se puede utilizar en la función filter(), como se ilustra en la Tabla 3.1:
| Symbol | Name |
|---|---|
== |
Igual a |
!= |
Distinto a |
>, <
|
Mayor/menor que |
>=, <=
|
Mayor/menor que o igual |
&, |, !
|
Operadores lógicos: Y, O, No (And, Or, Not) |
3.2.2 Encadenamiento de comandos con el operador pipe
Una de las claves para el flujo de trabajo con las funciones de dplyr es el operador ‘pipe’ %>% (y desde R 4.1.0 el pipe nativo |>), el cual toma su nombre del pipe de Unix | (Grolemund and Wickham 2016).
Los pipes permiten un código expresivo: el resultado de una función anterior se convierte en el primer argumento de la siguiente función, lo que permite encadenar.
Esto se ilustra a continuación, en el que sólo se filtran los países de Asia del conjunto de datos world, a continuación se seleccionan dos columnas para crear un subconjunto (name_long y continent) y las cinco primeras filas (resultado no mostrado).
world7 = world %>%
filter(continent == "Asia") %>%
dplyr::select(name_long, continent) %>%
slice(1:5)El fragmento anterior muestra cómo el operador pipe permite escribir los comandos en un orden claro:
los anteriores van de arriba a abajo (línea por línea) y de izquierda a derecha.
La alternativa a %>% son las llamadas a funciones ‘anidadas’, que son más difíciles de leer:
3.2.3 Agregación de atributos vectoriales
La agregación implica resumir los datos con una o más “variables de agrupación”, normalmente a partir de columnas del marco de datos que se va a agregar (la agregación geográfica se trata en el siguiente capítulo).
Un ejemplo de agregación de atributos es el cálculo del número de personas por continente a partir de los datos a nivel de país (una fila por país).
El conjunto de datos world contiene los ingredientes necesarios: las columnas pop y continent, la población y la variable de agrupación, respectivamente.
El objetivo es encontrar la suma sum() de las poblaciones de los países para cada continente, lo que resulta en un marco de datos más pequeño (la agregación es una forma de reducción de datos y puede ser un paso inicial útil cuando se trabaja con grandes conjuntos de datos).
Esto se puede hacer con la función básica de R agregate() de la siguiente manera:
world_agg1 = aggregate(pop ~ continent, FUN = sum, data = world, na.rm = TRUE)
class(world_agg1)
#> [1] "data.frame"El resultado es un marco de datos no espacial con seis filas, una por continente, y dos columnas que informan del nombre y la población de cada continente (véase la tabla 3.2 con los resultados de los 3 continentes más poblados).
aggregate() es una función genérica lo que significa que se comporta de forma diferente en función de lo que se le añada.
sf proporciona el método aggregate.sf() que se activa automáticamente cuando x es un objeto sf al que se le proporciona un argumento by:
world_agg2 = aggregate(world["pop"], list(world$continent), FUN = sum, na.rm = TRUE)
class(world_agg2)
#> [1] "sf" "data.frame"
nrow(world_agg2)
#> [1] 8El resultado es un objeto espacial world_agg2 que contiene 8 características que representan los continentes del mundo (y el océano abierto).
group_by() %>% summarize() es el equivalente en dplyr de la función aggregate(), con el nombre de la variable proporcionado en la función group_by() especificando la variable de agrupación y la información sobre lo que se va a resumir al pasarle la función summarize(), como se muestra a continuación:
El enfoque puede parecer más complejo, pero tiene ventajas: flexibilidad, legibilidad y control sobre los nuevos nombres de las columnas. Esta flexibilidad se ilustra en el siguiente comando, que calcula no sólo la población, sino también la superficie y el número de países de cada continente:
world_agg4 = world %>%
group_by(continent) %>%
summarize(pop = sum(pop, na.rm = TRUE), `area (sqkm)` = sum(area_km2), n = n())En el fragmento de código anterior pop, area (sqkm) y n son nombres de columnas en el resultado, y sum() y n() eran las funciones de agregación.
Estas funciones de agregación devuelven objetos sf con filas que representan los continentes y geometrías que contienen múltiples polígonos que representan cada masa de tierra e islas asociadas (esto funciona gracias a la operación geométrica “unión”, como se explica en la sección ??).
Combinemos lo que hemos aprendido hasta ahora sobre las funciones de dplyr, encadenando varios comandos para resumir datos de atributos sobre todos los países por continente.
El siguiente comando calcula la densidad de población (con mutate()), ordena los continentes por el número de países que contienen (con dplyr::arrange()), y mantiene sólo los 3 continentes más poblados (con top_n()), cuyo resultado se presenta en la Tabla 3.2):
world_agg5 = world %>%
st_drop_geometry() %>% # aisla la geometría para ganar velocidad
dplyr::select(pop, continent, area_km2) %>% # crea un subconjunto con las columnas de interés
group_by(continent) %>% # agrupa por continente y resume los datos
summarize(Pop = sum(pop, na.rm = TRUE), Area = sum(area_km2), N = n()) %>%
mutate(Density = round(Pop / Area)) %>% # calcula la densidad de población
top_n(n = 3, wt = Pop) %>% # muestra sólo las 3 primeras filas
arrange(desc(N)) # ordenar los 3 continentes en orden descendiente| continent | Pop | Area | N | Density |
|---|---|---|---|---|
| Africa | 1154946633 | 29946198 | 51 | 39 |
| Asia | 4311408059 | 31252459 | 47 | 138 |
| Europe | 669036256 | 23065219 | 39 | 29 |
?summarize y vignette(package = "dplyr") y en el capítulo 5 de R for Data Science.
3.2.4 Unión de atributos vectoriales
Combinar datos de diferentes fuentes es una tarea frecuente en la preparación de datos.
Las uniones hacen esto combinando tablas basadas en una variable “clave” compartida.
dplyr tiene múltiples funciones de unión incluyendo left_join() y inner_join() — véase vignette("two-table") para una lista completa.
Estos nombres de funciones siguen las convenciones utilizadas en el lenguaje de las bases de datos SQL (Grolemund and Wickham 2016, chap. 13); utilizándolos para unir conjuntos de datos no espaciales a objetos sf.
El objetivo de esta sección es aprender a realizar este tipo de unión.
Las funciones de unión de dplyr funcionan igual en los marcos de datos y en los objetos sf, la única diferencia importante es la columna de la lista geometría.
El resultado de las uniones de datos puede ser tanto un objeto sf como un objeto data.frame.
El tipo más común de unión de atributos en datos espaciales toma un objeto sf como primer argumento y le añade columnas de un data.frame especificado como segundo argumento.
Para demostrar las uniones, combinaremos los datos sobre la producción de café con el conjunto de datos world.
Los datos sobre el café se encuentran en un marco de datos llamado coffee_data del paquete spData (véase ?coffee_data para más detalles).
coffee_data tiene 3 columnas:
name_long nombra las principales naciones productoras de café; y coffee_production_2016 y coffee_production_2017 contienen valores estimados de la producción de café en unidades de sacos de 60 kg para cada año.
Un “left join”, que conserva el primer conjunto de datos, combina “world” con “coffee_data”:
world_coffee = left_join(world, coffee_data)
#> Joining, by = "name_long"
class(world_coffee)
#> [1] "sf" "tbl_df" "tbl" "data.frame"Dado que los conjuntos de datos de entrada comparten una “variable clave” (name_long), la unión ha funcionado sin utilizar el argumento by (véase ?left_join para más detalles).
El resultado es un objeto sf idéntico al objeto original world pero con dos nuevas variables sobre la producción de café.
Esto puede ser representado como un mapa, tal y como se ilustra en la Figura 3.1, generada con la función plot() a continuación:
names(world_coffee)
#> [1] "iso_a2" "name_long" "continent"
#> [4] "region_un" "subregion" "type"
#> [7] "area_km2" "pop" "lifeExp"
#> [10] "gdpPercap" "geom" "coffee_production_2016"
#> [13] "coffee_production_2017"
plot(world_coffee["coffee_production_2017"])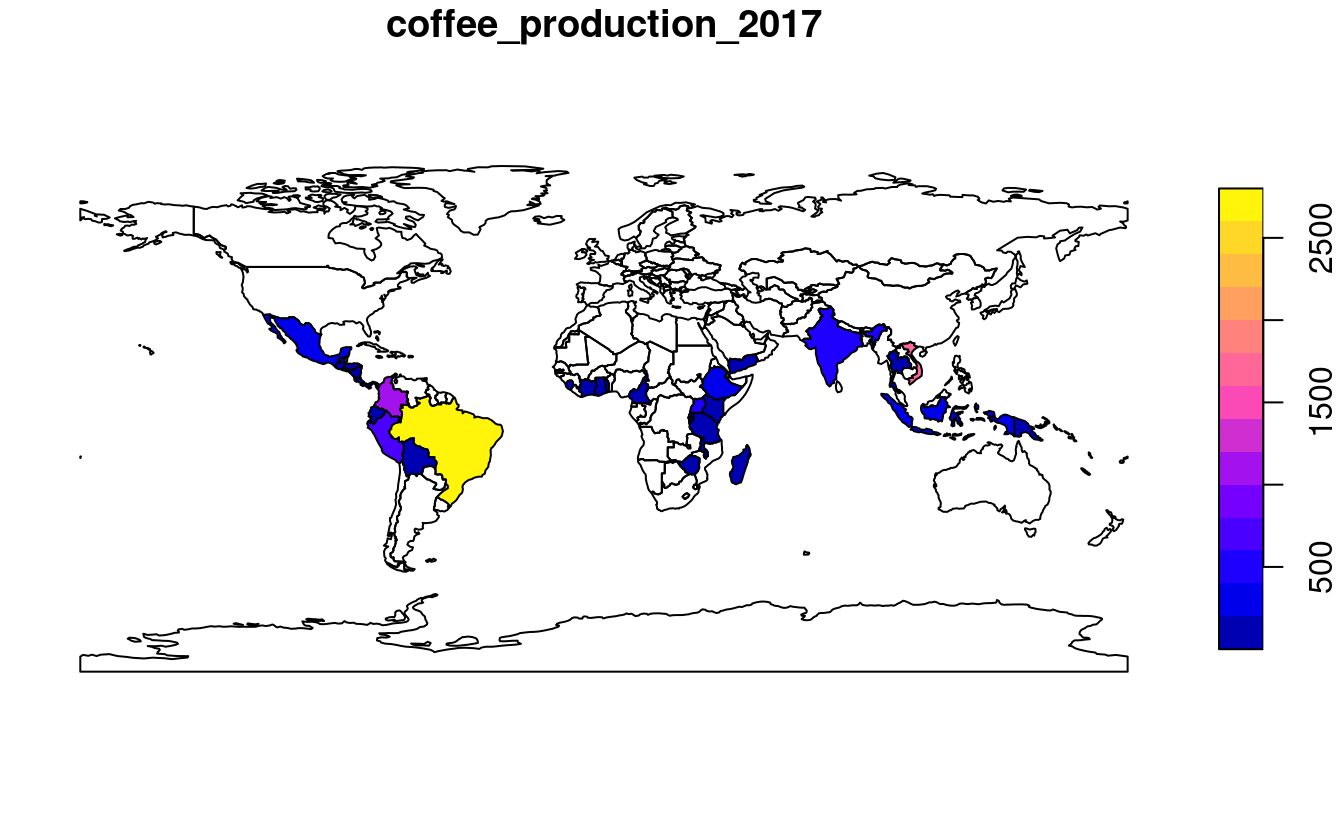
FIGURE 3.1: Producción mundial de café (en miles de sacos de 60 kg) por país, 2017. Fuente: Organización Internacional del Café.
Para que la unión funcione, se debe proporcionar una “variable clave” en ambos conjuntos de datos.
Por defecto dplyr utiliza todas las variables con nombres coincidentes.
En este caso, ambos objetos world_coffee y world contenían una variable llamada name_long, lo que explica el mensaje Joining, by = "name_long".
En la mayoría de los casos en que los nombres de las variables no son iguales. En esos casos tienes dos opciones:
- Cambiar el nombre de la variable clave en uno de los objetos para que coincidan.
- Utilizar el argumento
bypara especificar las variables de unión.
Este último enfoque se demuestra a continuación en una versión renombrada de coffee_data:
coffee_renamed = rename(coffee_data, nm = name_long)
world_coffee2 = left_join(world, coffee_renamed, by = c(name_long = "nm"))Obsérvese que se mantiene el nombre del objeto original, lo que significa que world_coffee y el nuevo objeto world_coffee2 son idénticos.
Otra característica del resultado es que tiene el mismo número de filas que el conjunto de datos original.
Aunque sólo hay 47 filas de datos en coffee_data, los 177 registros de países se mantienen intactos en world_coffee y world_coffee2:
Las filas del conjunto de datos original que no coinciden se les asignan valores “NA” para las nuevas variables de producción de café.
¿Y si sólo queremos conservar los países que coinciden con la variable clave?
En ese caso se puede utilizar inner join():
world_coffee_inner = inner_join(world, coffee_data)
#> Joining, by = "name_long"
nrow(world_coffee_inner)
#> [1] 45Fíjate en que el resultado de inner_join() sólo tiene 45 filas frente a las 47 de coffee_data.
¿Qué ha ocurrido con las filas restantes?
Podemos identificar las filas que no coinciden utilizando la función setdiff() de la siguiente manera:
setdiff(coffee_data$name_long, world$name_long)
#> [1] "Congo, Dem. Rep. of" "Others"El resultado muestra que Others representa una fila que no está presente en el conjunto de datos world y en el caso de la “República Democrática del Congo” el nombre
ha sido abreviado, lo que hace que la unión no lo tenga en cuenta.
El siguiente comando utiliza una función de concordancia de palabras (regex) del paquete stringr para confirmar qué debería ser Congo, Dem. Rep. of:
(drc = stringr::str_subset(world$name_long, "Dem*.+Congo"))
#> [1] "Democratic Republic of the Congo"Para solucionar este problema, crearemos una nueva versión de coffee_data y actualizaremos el nombre.
Si se une el marco de datos actualizado con inner_join(), se obtiene un resultado con las 46 naciones productoras de café:
coffee_data$name_long[grepl("Congo,", coffee_data$name_long)] = drc
world_coffee_match = inner_join(world, coffee_data)
#> Joining, by = "name_long"
nrow(world_coffee_match)
#> [1] 46También es posible unir en la dirección contraria: empezar con un conjunto de datos no espaciales y añadir variables de un objeto Simple Features (sf).
Esto se demuestra a continuación, se comienza con el objeto coffee_data y se le añaden variables del conjunto de datos original world.
A diferencia de las uniones anteriores, el resultado no es otro objeto Simple Feature, sino un marco de datos en forma de tibble de tidyverse:
El resultado de una unión tiende a coincidir con su primer argumento:
coffee_world = left_join(coffee_data, world)
#> Joining, by = "name_long"
class(coffee_world)
#> [1] "tbl_df" "tbl" "data.frame"sf.
La columna de geometría sólo puede utilizarse para crear mapas y operaciones espaciales si R “sabe” que es un objeto espacial, definido por un paquete espacial como sf.
Afortunadamente, los marcos de datos no espaciales con una columna de lista de geometría (como coffee_world) pueden ser convertidos en un objeto sf de la siguiente manera: st_as_sf(coffee_world).
Esta sección cubre la mayoría de los casos de uso de join.
Para más información, recomendamos Grolemund and Wickham (2016), la ‘viñeta’ de join en el paquete geocompkg que acompaña este libro, y la documentación del paquete data.table.11
Otro tipo de unión es la unión espacial, que se trata en el siguiente capítulo (Sección ??).
3.2.5 Creando atributos y eliminando información espacial
A menudo, queremos crear una nueva columna basada en columnas ya existentes.
Por ejemplo, queremos calcular la densidad de población de cada país.
Para ello necesitamos dividir una columna de población, aquí pop, por una columna de área, aquí area_km2.
Usando R base, podemos escribir:
world_new = world # no sobreescribe nuestros datos originales
world_new$pop_dens = world_new$pop / world_new$area_km2Alternativamente, podemos utilizar una de las funciones de dplyr - mutate() o transmute().
La función mutate() añade nuevas columnas en la penúltima posición del objeto sf (la última se reserva para la geometría):
La diferencia entre mutate() y transmute() es que esta última elimina todas las demás columnas existentes (excepto la columna de geometría fijada):
La función unite() del paquete tidyr (que proporciona muchas funciones útiles para remodelar conjuntos de datos, como pivot_longer()) pega las columnas existentes.
Por ejemplo, queremos combinar las columnas continent y region_un en una nueva columna llamada con_reg.
Además, podemos definir un separador (aquí: dos puntos :) que define cómo se deben unir los valores de las columnas de entrada, y si se deben eliminar las columnas originales (aquí: TRUE):
world_unite = world %>%
unite("con_reg", continent:region_un, sep = ":", remove = TRUE)La función separate() hace lo contrario de unite(): divide una columna en varias columnas utilizando una expresión regular o posiciones de caracteres.
Esta función también proviene del paquete tidyr.
La función de dplyr rename() y la función base de R setNames() son útiles para renombrar columnas.
La primera sustituye un nombre antiguo por uno nuevo.
El siguiente comando, por ejemplo, renombra la larga columna nombre_long a simplemente name:
setNames() cambia todos los nombres de las columnas a la vez, y requiere un vector de caracteres con un nombre que coincida con cada columna.
Esto se ilustra a continuación, lo cual produce el mismo objeto world, pero con nombres mucho más cortos:
new_names = c("i", "n", "c", "r", "s", "t", "a", "p", "l", "gP", "geom")
world %>%
setNames(new_names)Es importante señalar que las operaciones de datos de atributos conservan la geometría de simple features.
Como se ha mencionado al principio del capítulo, puede ser útil eliminar la geometría.
Para ello, hay que eliminarla explícitamente.
Por lo tanto, un enfoque como select(world, -geom) no tendrá éxito y en su lugar debe utilizar st_drop_geometry().12
world_data = world %>% st_drop_geometry()
class(world_data)
#> [1] "tbl_df" "tbl" "data.frame"3.3 Manipulando objetos raster
A diferencia del modelo de datos vectoriales subyacente a simple features (que representa puntos, líneas y polígonos como entidades discretas en el espacio), los datos rasterizados representan superficies continuas. Esta sección muestra cómo funcionan los objetos raster, creándolos desde cero, basándose en la sección ??. Debido a su estructura única, el subconjunto y otras operaciones con conjuntos de datos raster funcionan de manera diferente, como se demuestra en la sección ??.
El siguiente código recrea el conjunto de datos raster utilizados en la sección 2.3.4, cuyo resultado se ilustra en la figura 3.2.
Esto demuestra cómo funciona la función rast() para crear un raster de ejemplo llamado elev (que representa elevaciones).
elev = rast(nrows = 6, ncols = 6, resolution = 0.5,
xmin = -1.5, xmax = 1.5, ymin = -1.5, ymax = 1.5,
vals = 1:36)El resultado es un objeto raster con 6 filas y 6 columnas (especificadas por los argumentos nrow y ncol), y una extensión espacial mínima y máxima en dirección x e y (xmin, xmax, ymin, ymax).
El argumento vals establece los valores que contiene cada celda: datos numéricos que van de 1 a 36 en este caso.
Los objetos raster también pueden contener valores categóricos de clase lógica o variables factoriales en R.
El siguiente código crea un raster que representa el tamaño de los granos de café (Figura 3.2):
grain_order = c("clay", "silt", "sand")
grain_char = sample(grain_order, 36, replace = TRUE)
grain_fact = factor(grain_char, levels = grain_order)
grain = rast(nrows = 6, ncols = 6, resolution = 0.5,
xmin = -1.5, xmax = 1.5, ymin = -1.5, ymax = 1.5,
vals = grain_fact)El objeto raster almacena la correspondiente tabla de búsqueda o “Raster Attribute Table” (RAT) como una lista de marcos de datos, los cuales pueden ser visualizados con cats(grain) ( véase ?cats() para más información).
Cada elemento de esta lista es una capa del raster.
También es posible utilizar la función levels() para recuperar y añadir nuevos niveles de factores o sustituir los existentes:
levels(grain)[[1]] = c(levels(grain)[[1]], wetness = c("wet", "moist", "dry"))
#> Warning: [set.cats] setting categories like this is deprecated; use a two-column
#> data.frame instead
levels(grain)
#> [[1]]
#> value category
#> 1 0 0
#> 2 1 1
#> 3 2 2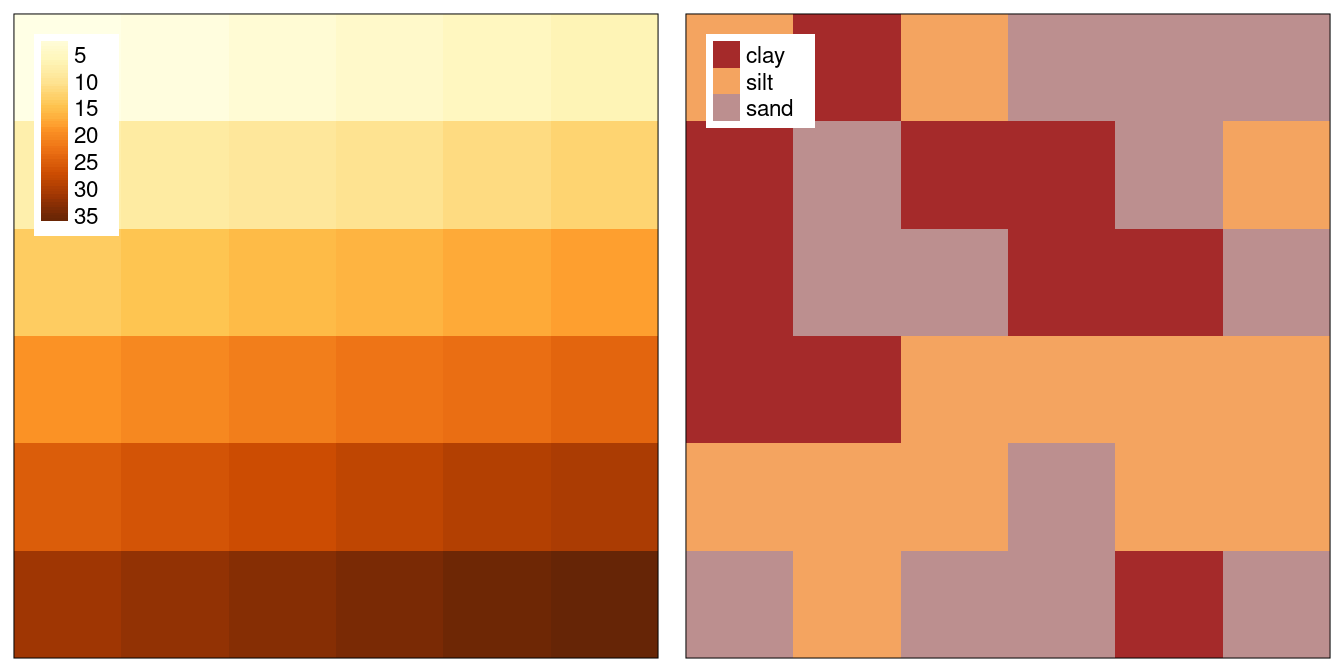
FIGURE 3.2: Conjuntos de datos rasterizados con valores numéricos (izquierda) y categóricos (derecha).
coltab() ( véase ?coltab).
Es importante señalar que al guardar un objeto rasterizado con una tabla de colores en un archivo (por ejemplo, GeoTIFF) también se guardará la información de color.
3.3.1 Subconjuntos de rásteres
Los subconjuntos de rásteres se realizan con el operador base de R [, que acepta varios tipos de entradas:
- Indexación de filas y columnas
- IDs de celdas
- Coordenadas (Véase la sección ??)
- Otros objetos espaciales (Véase la sección ??)
Aquí sólo mostramos las dos primeras opciones, ya que pueden considerarse operaciones no espaciales. Si necesitamos un objeto espacial para crear otro subconjunto o la salida es un objeto espacial, nos referimos a esto como subconjunto espacial. Por lo tanto, las dos últimas opciones se mostrarán en el próximo capítulo (véase la sección ??).
Las dos primeras opciones de subconjunto se demuestran en los comandos siguientes —
ambos devuelven el valor del píxel superior izquierdo en el objeto raster elev (los resultados no se muestran):
# fila 1, columna 1
elev[1, 1]
# ID de la celda 1
elev[1]El subconjunto de los objetos raster de varias capas devolverá el valor de la(s) celda(s) de cada capa.
Por ejemplo, c(elev, grain)[1] devuelve un marco de datos con una fila y dos columnas — una para cada capa.
Para extraer todos los valores o filas completas, también puedes utilizar values().
Los valores de las celdas pueden modificarse sobrescribiendo los valores existentes junto con una operación de subconjunto.
El siguiente fragmento de código, por ejemplo, establece la celda superior izquierda de elev a 0 (los resultados no se muestran):
elev[1, 1] = 0
elev[]Dejar los corchetes vacíos es una versión abreviada de values() para recuperar todos los valores de un raster.
También se pueden modificar múltiples celdas de esta manera:
elev[1, c(1, 2)] = 0Reemplazar los valores de rásteres multicapa puede hacerse con una matriz, con tantas columnas como capas queramos y filas como celdas reemplazables (resultados no mostrados):
3.3.2 Sintetizando objetos rasterizados
terra contiene funciones para extraer estadísticas descriptivas para rásteres enteros.
La impresión de un objeto raster en la consola al escribir su nombre devuelve sus valores mínimos y máximos.
summary() proporciona estadísticas descriptivas comunes. – mínimo, máximo, cuartiles y número de NAs para los rásteres continuos y un número de celdas de cada clase para los rásteres categóricos.
Otras operaciones de síntesis, como la desviación estándar (véase más adelante) o estadísticas de síntesis personalizadas, pueden calcularse con global().
global(elev, sd)summary() y global() a un objeto raster de varias capas, éstas resumirán cada capa por separado, como se puede ilustrar ejecutando: summary(c(elev, grain)).
Además, la función freq() permite obtener la tabla de frecuencias de los valores categóricos.
Las estadísticas de los valores raster pueden visualizarse de distintas maneras.
Funciones específicas como boxplot(), density(), hist() y pairs() funcionan también con objetos raster, como se demuestra en el histograma creado con el comando siguiente (no mostrado):
hist(elev)En caso de que la función de visualización deseada no funcione con objetos raster, se pueden extraer los datos raster para representarlos con la ayuda de values() (Sección ??).
Las estadísticas raster descriptivas pertenecen a las llamadas operaciones raster globales. Estas y otras operaciones típicas del procesamiento raster forman parte del esquema del álgebra de mapas, que se tratan en el siguiente capítulo (Sección ??).
Algunos nombres de funciones chocan entre paquetes (por ejemplo, una
función con el nombre extract() existe en ambos paquetes
terra y tidyr). Además de no cargar
los paquetes haciendo referencia a las funciones de forma verbosa (por
ejemplo, tidyr::extract()), otra forma de evitar los
choques de nombres de funciones es descargando el paquete que genere
este choque de nombres con detach(). El siguiente comando,
por ejemplo, descarga el paquete terra (esto también
puede hacerse en la pestaña paquete (Package) que reside por
defecto en el panel inferior derecho de RStudio):
detach(“paquete:terra”, unload = TRUE, force = TRUE). El
argumento force asegura que el paquete se desprenderá
incluso si otros paquetes dependen de él. Esto, sin embargo, puede
conducir a una usabilidad restringida de los paquetes que dependen del
paquete desprendido, y por lo tanto no se recomienda.
3.4 Ejercicios
Para estos ejercicios utilizaremos los conjuntos de datos us_states y us_states_df del paquete spData.
Antes de realizar estos ejercicios, deberás haber añadido este paquete y los otros utilizados en el capítulo de operaciones con atributos (sf, dplyr, terra) con comandos como library(spData).
us_states es un objeto espacial (de clase sf), que contiene la geometría y algunos atributos (incluyendo el nombre, la región, el área y la población) de los estados contiguos de Estados Unidos.
El objeto us_states_df es un marco de datos (de la clase data.frame) que contiene el nombre y variables adicionales (incluyendo la renta media y el nivel de pobreza, para los años 2010 y 2015) de los estados de EE.UU., incluyendo Alaska, Hawaii y Puerto Rico.
Los datos proceden de la Oficina del Censo de los Estados Unidos, y están documentados en “us_states” y “us_states_df”.
E1. Crea un nuevo objeto llamado us_states_name que contenga sólo la columna NAME del objeto us_states utilizando la sintaxis de R base ([) o tidyverse (select()).
¿Cuál es la clase del nuevo objeto y qué lo hace geográfico?
E2. Selecciona las columnas del objeto us_states que contienen datos de población.
Obtén el mismo resultado utilizando un comando diferente (bonus: intenta encontrar tres formas de obtener el mismo resultado).
Sugerencia: intenta utilizar funciones de ayuda, como contains o starts_with de dplyr (ver ?contains).
E3. Encuentra todos los estados con las siguientes características (bonus: encuéntralos y grafícalos):
- Pertenecen a la región del Medio Oeste.
- Pertenecen a la región Oeste, tienen una superficie inferior a 250.000 km2y en 2015 una población superior a 5.000.000 de habitantes (pista: puede que tengas que utilizar la función
units::set_units()oas.numeric()). - Pertenecen a la región Sur, tienen una superficie superior a 150.000 km2 o una población total en 2015 superior a 7.000.000 de residentes.
E4. ¿Cuál fue la población total en 2015 en el conjunto de datos us_states?
¿Cuál fue la población total mínima y máxima en 2015?
E5. ¿Cuántos estados hay en cada región?
E6. ¿Cuál fue la población total mínima y máxima en 2015 en cada región? ¿Cuál fue la población total en 2015 en cada región?
E7. Añade las variables de us_states_df a us_states, y crea un nuevo objeto llamado us_states_stats.
¿Qué función has utilizado y por qué?
¿Qué variable es la clave en ambos conjuntos de datos?
¿Cuál es la clase del nuevo objeto?
E8. us_states_df tiene dos filas más que us_states.
¿Cómo puedes encontrarlas? (pista: intenta utilizar la función dplyr::anti_join())
E9. ¿Cuál era la densidad de población en 2015 en cada estado? ¿Cuál era la densidad de población en cada estado en 2010?
E10. ¿Cuánto ha cambiado la densidad de población entre 2010 y 2015 en cada estado? Calcula el cambio en porcentajes y crea un mapa que lo muestre.
E11. Cambia los nombres de las columnas en us_states a minúsculas. (Sugerencia: las funciones - tolower() y colnames() pueden ayudar).
E12. Usando us_states y us_states_df crea un nuevo objeto llamado us_states_sel.
El nuevo objeto debe tener sólo dos variables - median_income_15 y geometry.
Cambia el nombre de la columna median_income_15 por Income.
E13. Calcula la variación del número de residentes que viven por debajo del nivel de pobreza entre 2010 y 2015 para cada estado. (Sugerencia: Consulta ?us_states_df para ver la documentación sobre las columnas relacionadas con el nivel de pobreza). Bonus: Calcula el cambio en el porcentaje de residentes que viven por debajo del nivel de pobreza en cada estado.
E14. ¿Cuál fue el número mínimo, medio y máximo de personas que viven por debajo del umbral de la pobreza en 2015 en cada región? Bonus: ¿Cuál es la región con el mayor aumento de personas que viven por debajo del umbral de la pobreza?
E15. Crea un raster desde cero con nueve filas y columnas y una resolución de 0,5 grados decimales (WGS84). Rellénalo con números aleatorios. Extrae los valores de las cuatro celdas de las esquinas.
E16. ¿Cuál es la clase más común de nuestro ejemplo de raster grain? (pista: modal())
E17. Traza el histograma y el boxplot del raster data(dem, package = "spDataLarge").