3 Opérations sur les tables attributaires
Prérequis
- Ce chapitre nécessite l’installation et le chargement des paquets suivant :
library(sf) # paquet pour les données vectorielles présenté dans le Chapitre 2
library(terra) # paquet pour les données raster présenté dans le Chapitre 2
library(dplyr) # paquet du tidyverse pour la manipulation de tableaux de données- Il s’appuie également sur spData, qui charge des jeux de données utilisés dans les exemples :
- Assurez-vous également d’avoir installé le paquet tidyr, ou le tidyverse dont il fait partie, si vous souhaitez exécuter des opérations restructurant des données pour la section 3.2.5.
3.1 Introduction
Les données attributaires sont des informations non spatiales associées à des données géographiques (géométrie).
Un arrêt de bus en est un exemple simple : sa position est généralement représentée par des coordonnées de latitude et de longitude (données géométriques), en plus de son nom.
L’arrêt Elephant & Castle / New Kent Road à Londres, par exemple, a pour coordonnées -0,098 degrés de longitude et 51,495 degrés de latitude, ce qui peut être représenté par POINT (-0,098 51,495) dans la représentation sfc décrite au chapitre 2.
Les attributs tels que le nom attribut de élément POINT (pour utiliser la terminologie de Simple Features) sont le sujet de ce chapitre.
Un autre exemple est la valeur d’altitude (attribut) pour un pixel spécifique dans les données raster. Contrairement au modèle de données vectorielles, le modèle de données raster stocke indirectement les coordonnées de la cellule de grille, ce qui signifie que la distinction entre attribut et information spatiale est moins claire. Pour illustrer ce point, pensez à un pixel dans la 3e ligne et la 4e colonne d’une matrice raster. Son emplacement spatial est défini par son indice dans la matrice : déplacez-vous depuis l’origine de quatre cellules dans la direction x (généralement vers l’est et la droite sur les cartes) et de trois cellules dans la direction y (généralement vers le sud et le bas). La résolution de la trame définit la distance pour chaque étape x et y qui est spécifiée dans l’en-tête du fichier. L’en-tête est un composant essentiel des ensembles de données raster qui spécifie comment les pixels se rapportent aux coordonnées géographiques (voir également le chapitre 4).
Vous apprendrez ainsi à manipuler des objets géographiques en fonction d’attributs, tels que le nom des arrêts de bus, dans un jeux de données vectorielles et l’altitude des pixels dans un jeux de données raster.
Pour les données vectorielles, cela implique des techniques telles que le sous-ensemble et l’agrégation (voir les sections ?? et ??).
Les sections ?? et 3.2.5 montrent respectivement comment joindre des données à des objets d’entités simples (simple features) à l’aide d’un ID (identifiant) partagé et comment créer de nouvelles variables.
Chacune de ces opérations a un équivalent spatial :
L’opérateur [ de la version de base de R, par exemple, fonctionne pour faire des sous-ensembles d’objets basé sur leur attribut que ces objets soient spatiaux ou non ; vous pouvez également joindre les attributs de deux jeux de données géographiques à l’aide de jointures spatiales.
C’est une bonne nouvelle : les compétences développées dans ce chapitre sont transférables.
Le chapitre 4 étend les méthodes présentées ici au monde spatial.
Après une exploration en profondeur dans les différents types d’opérations sur les attributs vectoriels, les opérations sur les données attributaires raster sont abordées dans la section ??. Elle montrera comment créer des couches raster contenant des attributs continus et catégoriels et comment extraire les valeurs des cellules d’une ou de plusieurs couches (sous-ensemble raster). La section ?? fournit une vue d’ensemble des calculs matricielles “globaux” qui peuvent être utilisées pour résumer des jeux de données raster entiers.
3.2 Manipulations des attributs de données vectorielles.
Les jeux de données géographiques vectorielles sont bien supportés dans R grâce à la classe sf, qui étend la classe data.frame de R.
Comme les tableaux de données, les objets sf ont une colonne par variable attributaire (comme le ‘nom’) et une ligne par observation ou entité (par exemple, par station de bus).
Les objets sf diffèrent des cadres de données de base parce qu’ils ont une colonne geometry de la classe sfc qui peut contenir une gamme d’entités géographiques (points simples et ‘multi’, lignes et polygones) par ligne.
Ceci a été décrit dans le chapitre 2, qui a démontré comment les méthodes génériques telles que plot() et summary() fonctionnent avec les objets sf.
sf fournit également des méthodes génériques permettant aux objets sf de se comporter comme des tableaux de données ordinaires, comme le montre l’impression des méthodes de la classe :
methods(class = "sf") # liste des 12 premières méthodes possibles avec la class sf
#> [1] aggregate cbind coerce
#> [4] initialize merge plot
#> [7] print rbind [
#> [10] [[<- $<- show Beaucoup d’entre elles (aggregate(), cbind(), merge(), rbind() et [) servent à manipuler des tableaux de données.
rbind(), par exemple, lie deux tableaux de données, l’un “au-dessus” de l’autre.
$<- crée de nouvelles colonnes.
Une caractéristique essentielle des objets sf est qu’ils stockent des données spatiales et non spatiales de la même manière, comme des colonnes dans un data.frame.
La colonne géométrique des objets sf est typiquement appelée geometry ou geom mais n´importe quel nom peut être utilisé.
La commande suivante, par exemple, crée une colonne géométrique nommée g :
st_sf(data.frame(n = world$name_long), g = world$geom)
wkb_geometry et the_geom.
Les objets sf peuvent également étendre les classes tidyverse pour les tableaux de données, tibble et tbl.
.
Ainsi, sf permet d’utiliser toute la puissance des capacités d’analyse de données de R sur les données géographiques, que vous utilisiez les fonctions de base de R ou du tidyverse pour l’analyse des données.
Les objets sf peuvent également être utilisés avec le package de traitement de données à hautes performances data.table bien que, comme documenté dans cette note Rdatatable/data.table#2273, ce n’est pas entièrement compatible avec les objets sf.
Avant d’utiliser ces capacités, il est utile de rappeler comment découvrir les propriétés de base des objets de données vectorielles.
Commençons par utiliser les fonctions de base de R pour découvrir l’ensemble de données world du paquet spData.
class(world) # c'est un objet sf et un (tidy) data.frame
#> [1] "sf" "tbl_df" "tbl" "data.frame"
dim(world) # c'est un objet de deux dimensions avec 177 lignes et 11 colonnes
#> [1] 177 11world contient dix colonnes non-géographiques (et une colonne de liste de géométrie) avec presque 200 lignes représentant les pays du monde.
La fonction st_drop_geometry() ne conserve que les données attributaires d’un objet sf, c’est-à-dire qu’elle supprime sa géométrie.
world_df = st_drop_geometry(world)
class(world_df)
#> [1] "tbl_df" "tbl" "data.frame"
ncol(world_df)
#> [1] 10Il peut être utile de supprimer la colonne de géométrie avant de travailler avec des données attributaires ; les processus de manipulation des données peuvent s’exécuter plus rapidement lorsqu’ils ne travaillent que sur les attributs et les colonnes de géométrie ne sont pas toujours nécessaires.
Dans la plupart des cas, cependant, il est judicieux de conserver la colonne géométrique, ce qui explique pourquoi cette colonne est “collante” (elle reste après la plupart des opérations sur les attributs, sauf si elle est spécifiquement abandonnée).
Les opérations de données non spatiales sur les objets sf ne modifient la géométrie d’un objet que lorsque cela est approprié (par exemple, en supprimant les frontières entre les polygones adjacents après l’agrégation).
Devenir compétent dans la manipulation des données d’attributs géographiques signifie devenir compétent dans la manipulation des tableaux de données.
Pour de nombreuses applications, le paquet du tidyverse dplyr offre une approche efficace pour travailler avec des tableaux de données.
La compatibilité avec le tidyverse est un avantage de sf par rapport à son prédécesseur sp, mais il y a quelques pièges à éviter (voir la vignette supplémentaire tidyverse-pitfalls à geocompr.github.io pour plus de détails).
3.2.1 Sélection de sous-ensemble dans des attributs de données vectorielles
Les méthodes de sélection de sous-ensembles de base de R incluent l’opérateur [ et la fonction subset().
Les principales fonctions de sélection de sous-ensembles dplyr sont filter() et slice() pour la sélection des lignes, et select() pour la sélection des colonnes.
Ces deux approches préservent les composantes spatiales des données attributaires dans les objets sf, tandis que l’utilisation de l’opérateur $ ou de la fonction dplyr pull() retournera une seule colonne d’attribut sous forme de vecteur et perdra les données attributaires, comme nous le verrons plus loin.
Cette section se concentre sur la sélection de sous-ensembles de tableaux de données sf ; pour plus de détails sur les cas de vecteurs et de tableaux de données non géographiques, nous vous recommandons de lire respectivement la section 2.7 de An Introduction to R (R Core Team, Smith, and Team 2021) et le chapitre 4 de Advanced R Programming (Wickham 2019).
L’opérateur [ peut sélectionner à la fois les lignes et les colonnes.
Il est possible de spécifier les éléments à conserver en indiquant leur rang entre crochets, directement après le nom de l’objet de type tableau de données qui les contient.
La commande object[i, j] signifie ’retourner les lignes représentées par i et les colonnes représentées par j, où i et j contiennent typiquement des entiers ou des TRUE et FALSE (les index peuvent aussi être des chaînes de caractères, indiquant les noms de lignes ou de colonnes).
Par exemple, objet[5, 1:3] signifie ’retourner des données contenant la cinquième ligne et les colonnes 1 à 3 : le résultat devrait être un tableau de données avec seulement une ligne et trois colonnes, et une quatrième colonne de géométrie si c’est un objet sf.
Laisser i ou j vide retourne toutes les lignes ou colonnes, donc world[1:5, ] retourne les cinq premières lignes et les 11 colonnes.
Les exemples ci-dessous illustrent les sélections avec cette syntaxe de base de R.
Devinez le nombre de lignes et de colonnes dans les tableaux de données sf retournés par chaque commande et vérifiez les résultats sur votre propre ordinateur (cf. la fin du chapitre pour d’autres exercices) :
world[1:6, ] # sélection de lignes par position
world[, 1:3] # sélection de colonnes par position
world[1:6, 1:3] # sélection de lignes et colonnes par position
world[, c("name_long", "pop")] # sélection de colonnes par leurs noms
world[, c(T, T, F, F, F, F, F, T, T, F, F)] # sélection en utilisant un vecteur logique
world[, 888] # un index référençant une colonne non-existanteUne démonstration de l’intérêt de l’utilisation de vecteurs logiques pour la sélection est démontrée dans le morceau de code ci-dessous.
Il crée un nouvel objet, small_countries, contenant les nations dont la surface est inférieure à 10,000 km2 :
i_small = world$area_km2 < 10000
summary(i_small) # on confirme le vecteur logique
#> Mode FALSE TRUE
#> logical 170 7
small_countries = world[i_small, ]L’objet intermédiaire i_small (variable indicatrice des petits pays) est un vecteur logique qui peut être utilisé pour sélectionner les sept plus petits pays du monde en fonction de leur superficie.
Une commande plus concise, qui omet l’objet intermédiaire, génère le même résultat :
small_countries = world[world$area_km2 < 10000, ]La fonction de base de R subset() fournit un autre moyen d’obtenir le même résultat :
small_countries = subset(world, area_km2 < 10000)Les fonctions de base de R sont matures, stables et largement utilisées, ce qui en fait un choix solide, en particulier dans les contextes où la reproductibilité et la fiabilité sont essentielles.
Les fonctions dplyr permettent des flux de travail “ordonnés” que certaines personnes (dont les auteurs de ce livre) trouvent intuitifs et productifs pour l’analyse interactive des données, en particulier lorsqu’elles sont associées à des éditeurs de code tels que RStudio qui permettent l’auto-complétion des noms de colonnes.
Les fonctions clés pour les sélection dans les tableaux de données (y compris les tableaux de données sf) avec les fonctions dplyr sont démontrées ci-dessous.
select() sélectionne les colonnes par nom ou par leur position.
Par exemple, vous pouvez sélectionner seulement deux colonnes, name_long et pop, avec la commande suivante :
Note : comme avec la commande équivalente de base de R (world[, c("name_long", "pop")]), la colonne collante geom reste.
select() permet également de sélectionner une plage de colonnes à l’aide de l’opérateur : :
# toutes les colonnes entre name_long et pop incluses
world2 = dplyr::select(world, name_long:pop)Vous pouvez supprimer des colonnes spécifiques avec l’opérateur - :
# toutes les colonnes sauf subregion et area_km2
world3 = dplyr::select(world, -subregion, -area_km2)Sélectionner et renommer les colonnes en même temps avec la syntaxe nouveau_nom = ancien_nom :
world4 = dplyr::select(world, name_long, population = pop)Il est intéressant de noter que la commande ci-dessus est plus concise que l’équivalent de base R, qui nécessite deux lignes de code :
world5 = world[, c("name_long", "pop")] # sélectionne les colonnes par nom
names(world5)[names(world5) == "pop"] = "population" # renommer les colonnesselect() fonctionne également avec des “fonctions d’aide” pour des opérations de sélections plus avancées, notamment contains(), starts_with() et num_range() (voir la page d’aide de ?select pour plus de détails).
La plupart des verbes de dplyr retournent un tableau de données, mais vous pouvez extraire une seule colonne comme un vecteur avec pull().
Vous pouvez obtenir le même résultat via la syntaxe de base de R avec les opérateurs de sélection de listes $ et [[, les trois commandes suivantes retournent le même tableau numérique :
pull(world, pop)
world$pop
world[["pop"]]slice() est l’équivalent pour les lignes de select().
Le morceau de code suivant, par exemple, sélectionne les lignes 1 à 6 :
slice(world, 1:6)filter() est l’équivalent pourdplyr de la fonction subset() de R de base.
Elle ne conserve que les lignes correspondant à des critères donnés, par exemple, uniquement les pays dont la superficie est inférieure à un certain seuil, ou dont l’espérance de vie moyenne est élevée, comme le montrent les exemples suivants :
world7 = filter(world ,area_km2 < 10000) # les pays avec une petite surface
world7 = filter(world, lifeExp > 82) # ceux avec une grande espérance de vieL’ensemble des opérateurs de comparaison peut être utilisé dans la fonction filter(), comme illustré dans le tableau 3.1 :
| Symbole | Nom |
|---|---|
== |
Egal à |
!= |
Non égal à |
>, <
|
Supérieur/inférieur à |
>=, <=
|
Supérieur/inférieur ou égal |
&, |, !
|
Opérateurs logiques : Et, Ou, Non |
3.2.2 Enchainement de commandes avec des pipes
La clé des flux de travail utilisant les fonctions dplyr est l’opérateur ‘pipe’ %>% (ou depuis R 4.1.0 le pipe natif |>), qui tire son nom du pipe Unix | (Grolemund and Wickham 2016).
Les pipes permettent un code expressif : la sortie d’une fonction précédente devient le premier argument de la fonction suivante, permettant l’enchaînement.
Ceci est illustré ci-dessous, dans lequel seuls les pays d’Asie sont filtrés de l’ensemble de données world, l’objet est ensuite sélectionné par les colonnes (name_long et continent) et les cinq premières lignes (résultat non montré).
Les lignes de code ci-dessus montrent comment l’opérateur pipe permet d’écrire des commandes dans un ordre précis :
les commandes ci-dessus sont écrites de haut en bas (ligne par ligne) et de gauche à droite.
L’alternative à %>% est un appel de fonction imbriqué, ce qui est plus difficile à lire :
Une autre alternative consiste à diviser les opérations en plusieurs lignes autonomes, ce qui est recommandé lors du développement de nouveaux paquets R, une approche qui a l’avantage de sauvegarder les résultats intermédiaires avec des noms distincts qui peuvent être inspectés ultérieurement à des fins de débogage (en contrepartie cette approche présente l’inconvénient d’être verbeuse et d’encombrer l’environnement global lors d’une analyse interactive) :
world9_filtered = filter(world, continent == "Asia")
world9_selected = dplyr::select(world9_filtered, continent)
world9 = slice(world9_selected, 1:5)Chaque approche présente des avantages et des inconvénients, dont l’importance dépend de votre style de programmation et de vos applications. Pour l’analyse interactive des données, fait l’objet de ce chapitre, nous trouvons que les opérations enchainées par des pipe sont rapides et intuitives, surtout lorsqu’elles sont combinées avec les raccourcis RStudio/VSCode pour créer des pipe et utiliser l’auto-complétion des noms de variables.
3.2.3 Agrégation sur la base des attributs de données vectorielles
L’agrégation consiste à résumer les données à l’aide d’une ou plusieurs “variables de regroupement”, généralement issues des colonnes du tableau de données à agréger (l’agrégation géographique est traitée au chapitre suivant).
Un exemple d’agrégation d’attributs est le calcul du nombre d’habitants par continent à partir des données par pays (une ligne par pays).
Le jeu de données world contient les ingrédients nécessaires : les colonnes pop et continent, respectivement la population et la variable de regroupement.
Le but est de trouver la somme() des populations des pays pour chaque continent, ce qui permet d’obtenir un tableau de données plus petit (l’agrégation est une forme de réduction des données et peut être une première étape utile lorsqu’on travaille avec de grands jeux de données).
Ceci peut être fait avec la fonction de base de R aggregate() comme suit :
world_agg1 = aggregate(pop ~ continent, FUN = sum, data = world,
na.rm = TRUE)
class(world_agg1)
#> [1] "data.frame"Le résultat est un tableau de données non spatialisées comportant six lignes, une par continent, et deux colonnes indiquant le nom et la population de chaque continent (voir le tableau 3.2 avec les résultats pour les 3 continents les plus peuplés).
aggregate() est une fonction générique ce qui signifie qu’elle se comporte différemment en fonction de ses entrées.
sf fournit la méthode aggregate.sf() qui est activée automatiquement lorsque x est un objet sf et qu’un argument by est fourni :
world_agg2 = aggregate(world["pop"], list(world$continent), FUN = sum,
na.rm = TRUE)
class(world_agg2)
#> [1] "sf" "data.frame"
nrow(world_agg2)
#> [1] 8L’objet world_agg2 résultant est un objet spatial contenant 8 entités représentant les continents du monde (et les océans).
group_by() |> summarize() est l’équivalent dplyr d’ aggregate(): le nom de variable indiqué dans la fonction group_by() spécifie la variable de regroupement et les informations sur ce qui doit être résumé sont passées à la fonction summarize(), comme indiqué ci-dessous :
Cette approche peut sembler plus complexe mais elle présente des avantages : flexibilité, lisibilité et contrôle sur les nouveaux noms de colonnes. Cette flexibilité est illustrée dans la commande ci-dessous, qui calcule non seulement la population mais aussi la superficie et le nombre de pays de chaque continent :
world_agg4 = world |>
group_by(continent) |>
summarize(pop = sum(pop, na.rm = TRUE), `area (sqkm)` = sum(area_km2), n = n())Dans le morceau de code précédent, pop, area (sqkm) et n sont des noms de colonnes du résultat, et sum() et n() sont les fonctions d’agrégation.
Ces fonctions d’agrégation renvoient des objets sf avec des lignes représentant les continents et des géométries contenant les multiples polygones représentant chaque masse terrestre et les îles associées (cela fonctionne grâce à l’opération géométrique ‘union’, comme expliqué dans la section ??).
Combinons ce que nous avons appris jusqu’à présent sur les fonctions dplyr, en enchaînant plusieurs commandes pour résumer les données attributaires des pays du monde entier par continent.
La commande suivante calcule la densité de population (avec mutate()), classe les continents selon le nombre de pays qu’ils contiennent (avec dplyr::arrange()), et ne conserve que les 3 continents les plus peuplés (avec dplyr::slice_max()), dont le résultat est présenté dans le Tableau 3.2) :
world_agg5 = world |>
st_drop_geometry() |> # enlève la colonne géométrie pour un gain de temps
dplyr::select(pop, continent, area_km2) %>% # sélectionne les colonnes d’intérêt
group_by(continent) |> # regroupe par continents et synthétise:
summarize(Pop = sum(pop, na.rm = TRUE), Superficie = sum(area_km2), N = n()) |>
mutate(Densité = round(Pop / Superficie)) |> # calcule la densité de population
slice_max(Pop, n = 3) |> # ne garde que les 3 plus peuplés
arrange(desc(N)) # trie par ordre du nombre de pays| continent | Pop | Superficie | N | Densité |
|---|---|---|---|---|
| Africa | 1154946633 | 29946198 | 51 | 39 |
| Asia | 4311408059 | 31252459 | 47 | 138 |
| Europe | 669036256 | 23065219 | 39 | 29 |
?summarize et vignette(package = "dplyr") et le chapitre 5 de R for Data Science.
3.2.4 Jointures attributaires de données vectorielles
La combinaison de données provenant de différentes sources est une tâche courante dans la préparation des données.
Les jointures le font en combinant des tables basées sur une variable “clé” partagée.
dplyr possède plusieurs fonctions de jointure, dont left_join() et inner_join() — voir vignette("two-table") pour une liste complète.
Ces noms de fonctions suivent les conventions utilisées dans le langage des base de données SQL (Grolemund and Wickham 2016, Chapitre 13) ; leur utilisation pour joindre des ensembles de données non spatiales à des objets sf est l’objet de cette section.
Les fonctions de jointure dplyr fonctionnent de la même manière sur les tableau de données et les objets sf, la seule différence importante étant la colonne de liste geometry.
Le résultat des jointures de données peut être un objet sf ou data.frame.
Le type le plus courant de jointure attributaures sur des données spatiales prend un objet sf comme premier argument et lui ajoute des colonnes à partir d’un data.frame spécifié comme second argument.
Pour découvrir les jointures, nous allons combiner les données sur la production de café avec l’ensemble de données world.
Les données sur le café sont dans un tableau de données appelé coffee_data du paquet spData (voir ?coffee_data pour plus de détails).
Il comporte 3 colonnes :
name_long nomme les principales nations productrices de café et coffee_production_2016 et coffee_production_2017 contiennent les valeurs estimées de la production de café en unités de sacs de 60 kg pour chaque année.
Une “jointure gauche” (left join), qui préserve le premier ensemble de données, fusionne world avec coffee_data :
world_coffee = left_join(world, coffee_data)
#> Joining with `by = join_by(name_long)`
class(world_coffee)
#> [1] "sf" "tbl_df" "tbl" "data.frame"Comme les données d’entrée partagent une “variable clé” (name_long), la jointure fonctionne sans utiliser l’argument by (voir ?left_join pour plus de détails).
Le résultat est un objet sf identique à l’objet original world mais avec deux nouvelles variables (indexées comme les colonne 11 et 12) sur la production de café.
Cet objet peut être représenté sous forme de carte, comme l’illustre la figure 3.1, générée avec la fonction plot() ci-dessous :
names(world_coffee)
#> [1] "iso_a2" "name_long" "continent"
#> [4] "region_un" "subregion" "type"
#> [7] "area_km2" "pop" "lifeExp"
#> [10] "gdpPercap" "geom" "coffee_production_2016"
#> [13] "coffee_production_2017"
plot(world_coffee["coffee_production_2017"])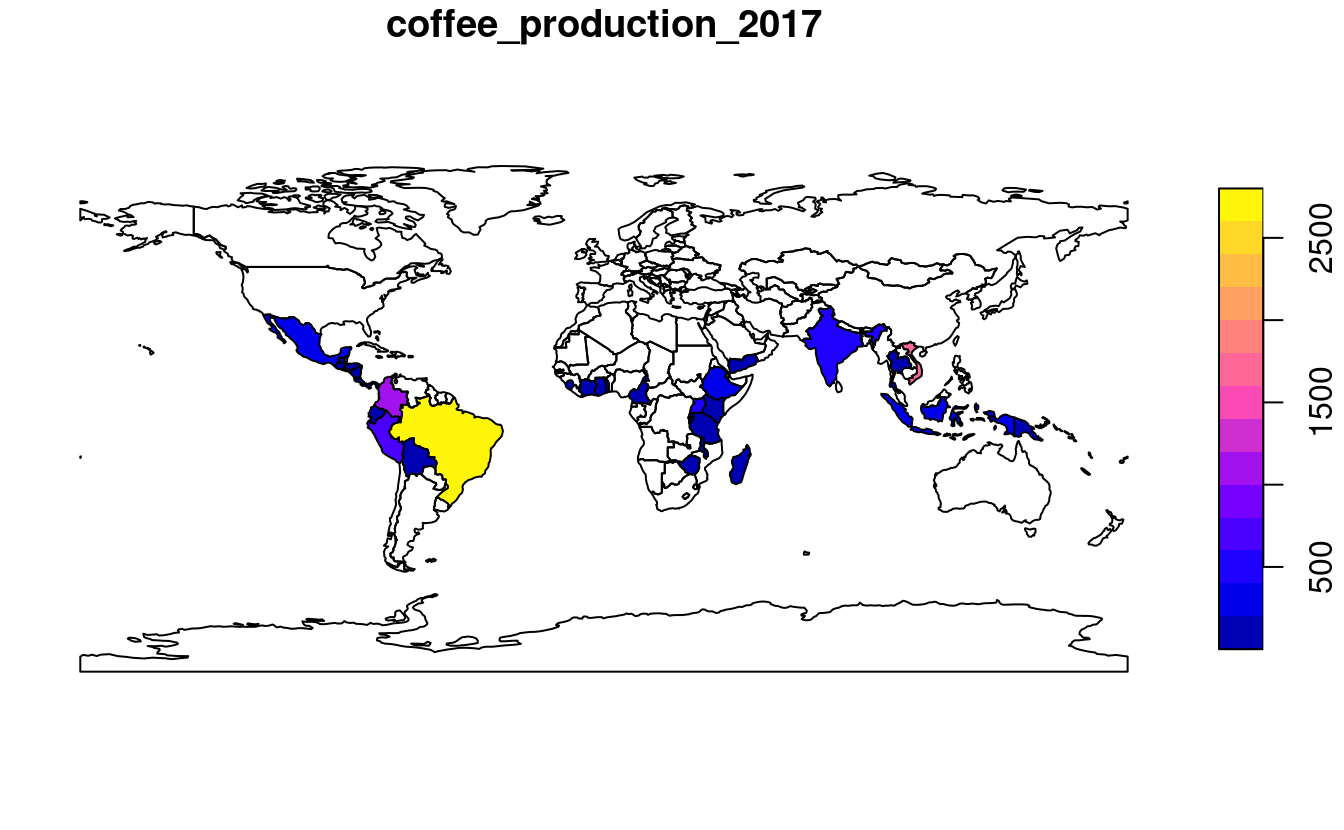
FIGURE 3.1: Production mondiale de café (milliers de sacs de 60 kg) par pays, 2017. Source : Organisation internationale du café.
Pour que la jointure fonctionne, une “variable clé” doit être fournie dans les deux ensembles de données.
Par défaut, dplyr utilise toutes les variables dont le nom correspond.
Dans ce cas, les deux objets world_coffee et world contenaient une variable appelée name_long, expliquant le message Joining, by = "name_long".
Dans la majorité des cas où les noms des variables ne sont pas les mêmes, vous avez deux options :
- Renommez la variable clé dans l’un des objets pour qu’ils correspondent.
- Utiliser l’argument
bypour spécifier les variables de jonction.
Cette dernière approche est présentée ci-dessous sur une version renommée de coffee_data :
coffee_renamed = rename(coffee_data, nm = name_long)
world_coffee2 = left_join(world, coffee_renamed, by = c(name_long = "nm"))Remarquez que la dénomination initiale est conservée, ce qui signifie que world_coffee et le nouvel objet world_coffee2 sont identiques.
Une autre caractéristique de l’objet final est qu’il a le même nombre de lignes que le jeu de données initial.
Bien qu’il n’y ait que 47 lignes de données dans coffee_data, les 177 enregistrements sont conservés tels quels dans world_coffee et world_coffee2:
les lignes du jeu de données initial pour lesquelles aucune correspondance n’est trouvée contiennent alors des valeurs NA pour les nouvelles variables relatives à la production de café.
Mais alors, comment procéder pour conserver uniquement les pays dont l’identifiant est présent dans les deux tables ?
Dans ce cas, il faut recourir à une jointure interne, ìnner join:
world_coffee_inner = inner_join(world, coffee_data)
#> Joining with `by = join_by(name_long)`
nrow(world_coffee_inner)
#> [1] 45Notez que le résultat de inner_join() ne comporte que 45 lignes contre 47 dans coffee_data.
Qu’est-il arrivé aux lignes restantes ?
Nous pouvons identifier les lignes qui ne correspondent pas en utilisant la fonction setdiff() comme suit :
setdiff(coffee_data$name_long, world$name_long)
#> [1] "Congo, Dem. Rep. of" "Others"Le résultat montre que “Others” représente une ligne non présente dans la base de données “monde” et que le nom de la “Democratic Republic of the Congo” représente l’autre ligne :
il a été abrégé, ce qui a fait que la jointure l’a manqué.
La commande suivante utilise une fonction de correspondance de chaîne (regex) du paquet stringr pour confirmer ce que devrait être Congo, Dem. Rep. of :
(drc = stringr::str_subset(world$name_long, "Dem*.+Congo"))
#> [1] "Democratic Republic of the Congo"Pour résoudre ce problème, nous allons créer une nouvelle version de coffee_data et mettre à jour le nom.
En utilisant le tableau de données mis à jour, inner_join() renvoie un résultat avec les 46 pays producteurs de café :
coffee_data$name_long[grepl("Congo,", coffee_data$name_long)] = drc
world_coffee_match = inner_join(world, coffee_data)
#> Joining with `by = join_by(name_long)`
nrow(world_coffee_match)
#> [1] 46Il est également possible d’effectuer une jointure dans l’autre sens : en partant d’un ensemble de données non spatiales et en ajoutant des variables provenant d’un objet spatial en entités simples (simples features).
Ci-dessous, on commence avec l’objet coffee_data et on ajoute les variables du jeux de données world.
Contrairement aux jointures précédentes, le résultat n’est pas un autre objet simple features , mais un tableau de données sous la forme d’un tibble tidyverse :
Le résultat d’une jointure tend à correspondre à son premier argument :
coffee_world = left_join(coffee_data, world)
#> Joining with `by = join_by(name_long)`
class(coffee_world)
#> [1] "tbl_df" "tbl" "data.frame"sf.
Elle ne peut être utilisée pour créer des cartes et des opérations spatiales que si R “sait” qu´il s´agit d´un objet spatial, défini par un paquet spatial tel que sf.
Heureusement, les tableaux de données non spatiaux avec une colonne de liste de géométrie (comme coffee_world) peuvent être convertis en un objet sf comme suit : st_as_sf(coffee_world).
Cette section couvre la majorité des cas d’utilisation de la jointure. Pour plus d’informations, nous recommandons la lecture du chapitre Données relationnelles dans Grolemund and Wickham (2016), la vignette join dans le paquet geocompkg qui accompagne ce livre, et la documentation décrivant les jointures avec data.table et d’autres paquets. Les jointures spatiales sont traitées dans le chapitre suivant (Section ??).
3.2.5 Création d’attributs et suppression d’informations spatiales
Souvent, nous souhaitons créer une nouvelle colonne à partir de colonnes déjà existantes.
Par exemple, nous voulons calculer la densité de population pour chaque pays.
Pour cela, nous devons diviser une colonne de population, ici pop, par une colonne de surface, ici area_km2 avec une unité de surface en kilomètres carrés.
En utilisant les fonctions de base de R, nous pouvons écrire :
world_new = world # afin de ne pas écraser nos données
world_new$pop_dens = world_new$pop / world_new$area_km2Alternativement, nous pouvons utiliser l’une des fonctions dplyr - mutate() ou transmute().
mutate() ajoute de nouvelles colonnes à l’avant-dernière position dans l’objet sf (la dernière est réservée à la géométrie) :
world |>
mutate(pop_dens = pop / area_km2)La différence entre mutate() et transmute() est que ce dernier supprime toutes les autres colonnes existantes (à l’exception de la colonne de géométrie collante) :
world |>
transmute(pop_dens = pop / area_km2)unite() du paquet tidyr (qui fournit de nombreuses fonctions utiles pour remodeler les ensembles de données, notamment pivot_longer()) colle ensemble des colonnes existantes.
Par exemple, ici nous voulons combiner les colonnes continent et region_un dans une nouvelle colonne nommée con_reg.
De plus, nous pouvons définir un séparateur (ici : deux points :) qui définit comment les valeurs des colonnes d’entrée doivent être jointes, et si les colonnes originales doivent être supprimées (ici : TRUE) :
world_unite = world |>
tidyr::unite("con_reg", continent:region_un, sep = ":", remove = TRUE)L’objet sf résultant a une nouvelle colonne appelée con_reg représentant le continent et la région de chaque pays, par exemple South America:Americas pour l’Argentine et les autres pays d’Amérique du Sud.
La fonction separate() de tidyr fait l’inverse de unite() : elle divise une colonne en plusieurs en utilisant soit une expression régulière, soit la position des caractères.
La fonction dplyr rename() et la fonction de base de R setNames() sont utiles pour renommer des colonnes.
La première remplace un ancien nom par un nouveau.
Ici, par exemple, elle renomme la longue colonne name_long en un simple name :
world |>
rename(name = name_long)setNames() change tous les noms de colonnes en une fois, et nécessite un vecteur de caractères avec un nom correspondant pour chaque colonne.
Ceci est illustré ci-dessous et produit le même objet world mais avec des noms très courts :
new_names = c("i", "n", "c", "r", "s", "t", "a", "p", "l", "gP", "geom")
world_new_names = world |>
setNames(new_names)Il est important de noter que les opérations sur les données attributaires préservent la géométrie des entités simples.
Parfois, par exemple pour rendre un processus plus rapide, il peut être utile de supprimer la géométrie.
Il faut utiliser pour cela st_drop_geometry(), et non le faire manuellement avec des commandes telles que select(world, -geom), comme montré ci-dessous . 20
world_data = world |> st_drop_geometry()
class(world_data)
#> [1] "tbl_df" "tbl" "data.frame"3.3 Manipuler des objets raster
Contrairement au modèle de données vectorielles sous-tendu par les entités simples (qui représente les points, les lignes et les polygones comme des entités discrètes dans l’espace), les données matricielles représentent des surfaces continues. Cette section présente le fonctionnement des objets raster en les créant de bout en bout, en s’appuyant sur la section ??. En raison de leur structure unique, les sélections et les autres opérations sur les jeux de données raster fonctionnent d’une manière différente, comme le montre la section ??.
Le code suivant recrée le jeu de données matricielles utilisé dans la section 2.3.4, dont le résultat est illustré dans la figure 3.2.
Cela montre comment la fonction rast() fonctionne pour créer un exemple de données matricielles nommé elev (représentant les altitudes).
elev = rast(nrows = 6, ncols = 6, resolution = 0.5,
xmin = -1.5, xmax = 1.5, ymin = -1.5, ymax = 1.5,
vals = 1:36)Le résultat est un objet raster avec 6 lignes et 6 colonnes (spécifiées par les arguments nrow et ncol), et une étendue spatiale comprise dans les directions x et y (xmin, xmax, ymin, ymax).
L’argument vals définit les valeurs que chaque cellule contient : des données numériques allant de 1 à 36 dans ce cas.
Les objets raster peuvent également contenir des valeurs catégorielles de la classe des variables logiques ou factor de R.
Le code suivant crée les ensembles de données matricielles illustrés dans la figure 3.2 :
grain_order = c("clay", "silt", "sand")
grain_char = sample(grain_order, 36, replace = TRUE)
grain_fact = factor(grain_char, levels = grain_order)
grain = rast(nrows = 6, ncols = 6, resolution = 0.5,
xmin = -1.5, xmax = 1.5, ymin = -1.5, ymax = 1.5,
vals = grain_fact)L’objet raster stocke la table de correspondance ou “Raster Attribute Table” (RAT) correspondante sous la forme d’une liste de tableaux de données, qui peuvent être visualisés avec cats(grain) (cf. ?cats() pour plus d’informations).
Chaque élément de cette liste est une couche du raster.
Il est également possible d’utiliser la fonction levels() pour récupérer et ajouter de nouveaux niveaux de facteurs ou remplacer des niveaux existants
levels(grain) = data.frame(value = c(0, 1, 2), wetness = c("wet", "moist", "dry"))
levels(grain)
#> [[1]]
#> value wetness
#> 1 0 wet
#> 2 1 moist
#> 3 2 dry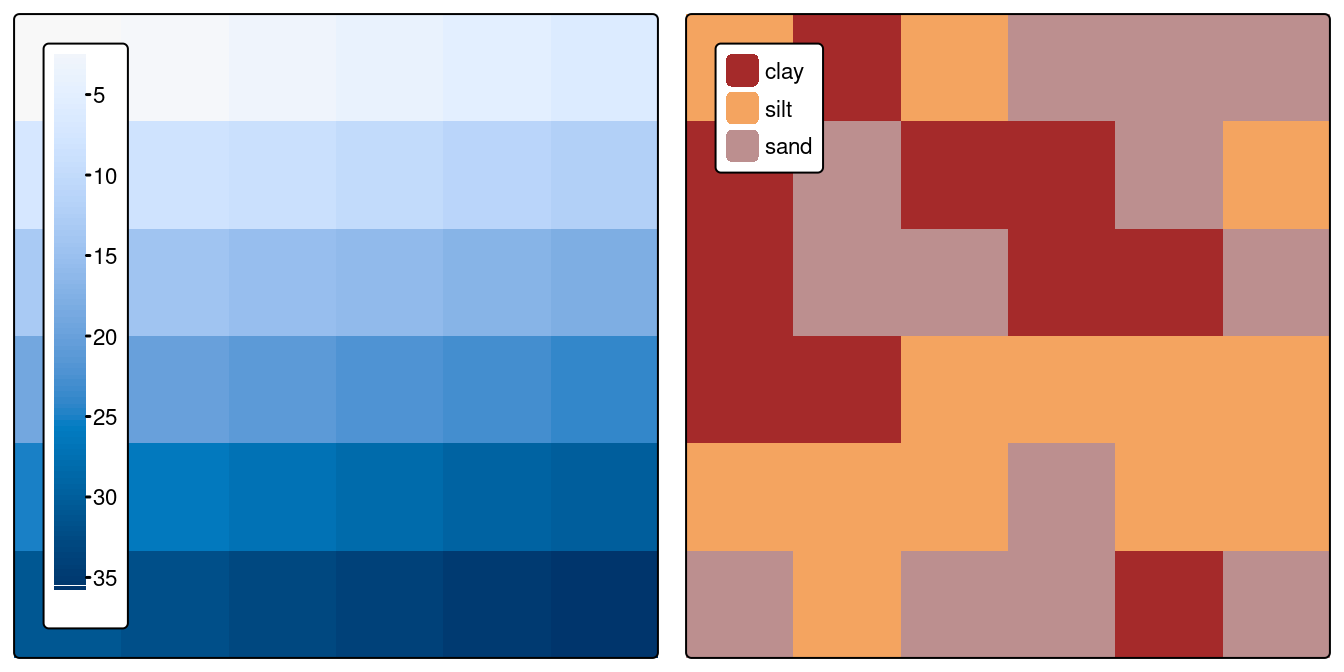
FIGURE 3.2: Jeux de données raster avec des valeurs numériques (à gauche) et catégorielles (à droite).
coltab() (voir ?coltab).
Il est important de noter que la sauvegarde d´un objet raster avec une table de couleurs dans un fichier (par exemple, GeoTIFF) sauvegardera également les informations de couleurs
3.3.1 Sélection sur des raster
La sélection de données raster est réalisée à l’aide de l’opérateur de base de R [, qui accepte une large gamme d’entrées :
- indexation ligne-colonne ;
- ID des cellules ;
- coordonnées (voir la section 4.3.1) ;
- autre objet spatial (voir la section 4.3.1).
Nous ne présentons ici que les deux premières options, car elles peuvent être considérées comme des opérations non spatiales. Si nous avons besoin d’un objet spatial pour en sélectionner un autre ou si le résultat est un objet spatial, nous en parlerons comme une sélection spatiale. Par conséquent, les deux dernières options seront présentées dans le chapitre suivant (voir la section 4.3.1).
Les deux premières options de sous-ensembles sont présentées dans les commandes ci-dessous —
toutes deux renvoient la valeur du pixel supérieur gauche dans l’objet raster elev (résultats non montrés) :
# ligne 1, colonne 1
elev[1, 1]
# pixel ID 1
elev[1]Les sélections d’objets raster à couches multiples renverront la ou les valeurs des cellules pour chaque couche.
Par exemple, two_layers = c(grain, elev); two_layers[1] renvoie un tableau de données avec une ligne et deux colonnes — une pour chaque couche.
Pour extraire toutes les valeurs ou des lignes complètes, vous pouvez également utiliser values().
Les valeurs des cellules peuvent être modifiées en écrasant les valeurs existantes en conjonction avec une opération de sélection.
L’exemple de code suivant, par exemple, définit la cellule supérieure gauche de elev à 0 (résultats non montrés) :
elev[1, 1] = 0
elev[]Laisser les crochets vides est une version raccourcie de values() pour récupérer toutes les valeurs d’un raster.
Plusieurs cellules peuvent également être modifiées de cette manière :
elev[1, c(1, 2)] = 0Le remplacement des valeurs des rasters multicouches peut se faire avec une matrice comportant autant de colonnes que de couches et de lignes que de cellules remplaçables (résultats non montrés) :
3.3.2 Résumer les objets raster
terra contient des fonctions permettant d’extraire des statistiques descriptives pour des rasters entiers.
L’impression d’un objet raster sur la console en tapant son nom renvoie les valeurs minimales et maximales d’un objet raster.
summary() fournit des statistiques descriptives courantes – minimum, maximum, quartiles et nombre de NAs pour les matrices continues et un nombre de cellules de chaque classe pour les matrices catégorielles.
D’autres opérations de synthèse telles que l’écart-type (voir ci-dessous) ou des statistiques de synthèse personnalisées peuvent être calculées avec global().
global(elev, sd)summary() et global() un objet raster multi-couches, elles résumeront chaque couche séparément, comme on peut l´illustrer en exécutant : summary(c(elev, grain)).
De plus, la fonction freq() permet d’obtenir le tableau de fréquence des valeurs catégorielles.
Les statistiques des données raster peuvent être visualisées de différentes manières.
Des fonctions spécifiques telles que boxplot(), density(), hist() et pairs() fonctionnent également avec les objets raster, comme le montre l’histogramme créé avec la commande ci-dessous (non montré) :
hist(elev)Si la fonction de visualisation souhaitée ne fonctionne pas avec les objets raster, on peut extraire les données à représenter à l’aide de values() (section ??).
Les statistiques descriptives sur des raster font partie des opérations raster dites globales. Ces opérations, ainsi que d’autres opérations de traitement raster typiques, font partie du traitement algébrique sur raster, qui est abordé dans le chapitre suivant (section ??).
Certains noms de fonctions sont réutilisés entre les paquets et
rentrent en conflit(par exemple, une fonction avec le nom
extract() existe à la fois dans les paquets
terra et tidyr). En plus de ne pas
charger les paquets en se référant verbalement aux fonctions (par
exemple, tidyr::extract()), une autre façon d’éviter les
conflits de noms de fonctions est de décharger le paquet en question
avec detach(). La commande suivante, par exemple, décharge
le paquet terra (ceci peut également être fait dans
l’onglet package qui se trouve par défaut dans le panneau
inférieur droit de RStudio) :
detach(“package:terra”, unload = TRUE, force = TRUE).
L’argument force permet de s’assurer que le paquet sera
détaché même si d’autres paquets en dépendent. Cependant, cela peut
conduire à une utilisation restreinte des paquets dépendant du paquet
détaché, et n’est donc pas recommandé.
3.4 Exercices
Pour ces exercices, nous allons utiliser les jeux de données us_states et us_states_df du paquetage spData.
Vous devez avoir chargé ce paquet, ainsi que les autres paquets utilisés dans le chapitre sur les opérations d’attributs (sf, dplyr, terra) avec des commandes telles que library(spData) avant de tenter ces exercices :
us_states est un objet spatial (de classe sf), contenant la géométrie et quelques attributs (dont le nom, la région, la superficie et la population) des états des États-Unis contigus.
us_states_df est un tableau de données (de classe data.frame) contenant le nom et des variables supplémentaires (dont le revenu médian et le niveau de pauvreté, pour les années 2010 et 2015) des états américains, y compris l’Alaska, Hawaii et Porto Rico.
Les données proviennent du United States Census Bureau, et sont documentées dans ?us_states et ?us_states_df.
E1. Créez un nouvel objet appelé us_states_name qui ne contient que la colonne NAME de l’objet us_states en utilisant la syntaxe de base de R ([) ou du tidyverse (select()).
Quelle est la classe du nouvel objet et qu’est-ce qui le rend géographique ?
E2. Sélectionnez les colonnes de l’objet us_states qui contiennent des données sur la population.
Obtenez le même résultat en utilisant une commande différente (bonus : essayez de trouver trois façons d’obtenir le même résultat).
Indice : essayez d’utiliser les fonctions d’aide, telles que contains ou matches du paquet dplyr (voir ?contains).
E3. Trouvez tous les États ayant les caractéristiques suivantes (bonus : trouvez et représentez-les) :
- Appartenir à la région du Midwest.
- Appartenir à la région Ouest, avoir une superficie inférieure à 250 000 km2et en 2015 une population supérieure à 5 000 000 de résidents (indice : vous devrez peut-être utiliser la fonction
units::set_units()ouas.numeric()). - Appartenant à la région Sud, avoir une superficie supérieure à 150 000 km2et une population totale en 2015 supérieure à 7 000 000 de résidents.
E4. Quelle était la population totale en 2015 dans l’ensemble de données us_states ?
Quel était le minimum et le maximum de la population totale en 2015 ?
E5. Combien d’États y a-t-il dans chaque région ?
E6. Quelle était la population totale minimale et maximale en 2015 dans chaque région ? Quelle était la population totale en 2015 dans chaque région ?
E7. Effectuez une jointure entre les variables de us_states_df à us_states, et créez un nouvel objet appelé us_states_stats.
Quelle fonction avez-vous utilisée et pourquoi ?
Quelle variable est la clé dans les deux ensembles de données ?
Quelle est la classe du nouvel objet ?
E8. us_states_df a deux lignes de plus que us_states.
Comment pouvez-vous les trouver ? (indice : essayez d’utiliser la fonction dplyr::anti_join())
E9. Quelle était la densité de la population en 2015 dans chaque État ? Quelle était la densité de la population en 2010 dans chaque État ?
E10. Estimez le changement de la densité de la population entre 2010 et 2015 dans chaque État. Calculez ce changement en pourcentages et cartographiez-les.
E11. Changez les noms des colonnes dans us_states en minuscules. (Indice : les fonctions d’aide - tolower() et colnames() peuvent vous aider).
E12. En utilisant us_states et us_states_df, créez un nouvel objet appelé us_states_sel.
Ce nouvel objet ne doit avoir que deux variables - median_income_15 et geometry.
Changez le nom de la colonne median_income_15 en Income.
E13. Calculez l’évolution du nombre de résidents vivant sous le seuil de pauvreté entre 2010 et 2015 pour chaque État. (Conseil : voir ?us_states_df pour la documentation sur les colonnes traitant du niveau de pauvreté). Bonus : Calculez l’évolution du pourcentage de résidents vivant sous le seuil de pauvreté dans chaque État.
E13. Calculez l’évolution du nombre de résidents vivant sous le seuil de pauvreté entre 2010 et 2015 pour chaque État. (Conseil : voir ?us_states_df pour la documentation sur les colonnes traitant du niveau de pauvreté). Bonus : Calculez l’évolution du pourcentage de résidents vivant sous le seuil de pauvreté dans chaque État.
E15. Créez un raster à partir de zéro avec neuf lignes et colonnes et une résolution de 0,5 degré décimal (WGS84). Remplissez-le de nombres aléatoires. Sélectionnez les valeurs des cellules de chaque coin.
E16. Quelle est la classe la plus commune de notre exemple de raster grain (indice : modal) ?
E17. Utilisez un histogramme et un boxplot sur le fichier dem.tif du paquet spDataLarge (system.file("raster/dem.tif", package = "spDataLarge")).